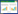中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 李高磊, 黄玮, 孙浩, 李余动. 2021
- Gaolei Li, Wei Huang, Hao Sun, Yudong Li. 2021
- 机器学习在微生物组宿主表型预测中的应用
- Applications of machine learning in predicting host phenotype based on microbiome
- 微生物学报, 61(9): 2581-2593
- Acta Microbiologica Sinica, 61(9): 2581-2593
-
文章历史
- 收稿日期:2020-10-10
- 修回日期:2021-03-08
- 网络出版日期:2021-03-26
人体微生物群(microbiota)是所有生活在人体内部和表面的微生物的集合,由多种微生物组成,包括细菌、古菌、真菌和病毒等,主要分布在口腔、皮肤、胃肠道和泌尿生殖道等部位。例如人类口腔中定殖了超过600种不同的细菌、病毒、真菌及衣原体等微生物物种[1]。这些微生物群的遗传信息的总和称为人类微生物组(microbiome),是人体本身基因组外的第二基因组(second genome)。随着人类微生物组计划的开展,越来越多的研究表明,人体微生物群落结构的变化与许多疾病相关,如炎症性肠病(IBD)、糖尿病、肥胖、肿瘤等,而菌群失调(dysbiosis)的干预手段将可用于治疗疾病[2]。因此,了解人体微生物组的多样性和分布,特别是在不同生理和疾病状态下发生的变化,或发现微生物群落组成与疾病临床特征之间的关联性,将有助于疾病的临床检测、诊断和治疗[3-4]。目前基于微生物组数据进行准确的疾病诊断与治疗已成为生物医学领域研究的热点方向之一。
随着基因测序技术的进步和测序成本不断下降,大样本量的微生物组学研究激增。传统的统计方法已经不再适用于极度高维、稀疏的微生物组数据分析,而适用于复杂数据分析的机器学习逐渐成为微生物组学数据分析的首选方法[5]。机器学习(machine learning,ML)让计算机能够自主“学习” (拟合训练数据),通过神经网络、决策树等算法,从数据中自动分析寻找规律,从而生成经验模型,用于新数据的预测或分类。机器学习算法的主要理论成果在20世纪60年代就已产生,伴随高性能计算和大数据技术的发展,又产生深度学习(deep learning,DL)等人工智能技术。深度学习能够很好地处理组学序列数据,自动确定关键特征(features)用于构建模型,从而对疾病进行准确的预测和诊断等[6]。目前机器学习已被广泛应用于生物医学与临床研究,例如DeepMind Health团队致力于开发有效的深度学习技术帮助医生鉴定有急性肾损伤风险的患者(https://github.com/LiLabZSU/microbioML)。欧盟于2019年启动了ML4Microbiome项目(https://www.ml4microbiome.eu/),旨在优化和规范机器学习在微生物组研究中的应用。
机器学习方法往往需要结合统计学、概率论、线性代数和算法复杂性理论等数学知识,对于生物医学研究者是一个巨大的挑战。本文综述了机器学习方法在基于微生物组数据预测宿主表型方面的应用,简要介绍了微生物组研究中常用的机器学习方法、数据处理步骤以及性能评价指标,并提供了一个基于唾液微生物组数据预测口腔异味的研究案例。
1 微生物组数据微生物组研究一般可分16S rRNA基因扩增子(amplicon)测序与全基因组鸟枪法(shotgun)测序两种策略。根据不同的研究目的,可从唾液、皮肤、粪便或血液等中收集微生物样本。微生物样品的16S rRNA基因测序数据需要使用微生物组分析流程(如QIIME或mothur等)处理,按序列相似度聚类为不同簇(cluster),即可操作分类单位(OTU),再用于后续微生物多样性分析。OTU聚类一般有3种方法:(1)通过直接与参考序列数据库进行序列比对来确定OTU (closed-reference);(2) 如果没有参考序列数据库,可从头开始聚类来获得OTU,这种是从头聚类法(de novo clustering);(3) 先基于参考序列来获得OTU聚类,剩下不能与参考序列匹配的序列,再重新进行从头聚类(open-reference)。一旦确定了OTU簇,就可以通过RDP Classifier等方法为每个OTU的代表序列指定物种分类信息。16S rRNA数据已经被证实具有较高分类准确性,关于从实验样品中获得微生物组序列的方法的更多细节可参见文献[7]。
OTU丰度数据是最常用的微生物群落与宿主性状关联分析的输入特征(features)。如表 1所示,OTU表为菌群计数数据的n×m矩阵X[n, m],其中n为OTU特征数,m为样本数。Y(m)为长度为m的宿主性状(traits)向量,性状可能是一个分类数据,如受试者有无口臭症状,或者是一个连续数据,如身体重量指数(BMI)。OTU表根据序列相似性聚类后得到的OTU簇数量表示特定分类单元的丰度,避免了只对相同序列进行分组而导致的过度稀疏。由于微生物组研究的样本数一般远少于OTU或物种数,造成数据集的维度较高,即每一个样本有很多特征。OTU表的高维特性使其难以用传统统计学方法进行数据处理,机器学习作为分析高维数据的一种有效手段,可以用于阐明微生物类群(或其他宏基因组特征)与宿主或环境属性之间的联系[8]。
| #OTU_ID | Sample1 | Sample2 | Sample3 | Sample4 | Sample5 |
| OTU3 | 18 | 10 | 166 | 16 | 66 |
| OTU7 | 29 | 15 | 350 | 35 | 50 |
| OTU35 | 29 | 24 | 112 | 17 | 92 |
| OTU78 | 76 | 57 | 0 | 62 | 0 |
| OTU115 | 22 | 36 | 182 | 0 | 0 |
| Trait | P | N | P | P | N |
| a Each row represents an OTU, and each column represents a sample. The last row represents a host trait (P stands for positive and N stands for negative). | |||||
2 机器学习算法
根据训练数据是否带有标签(tag),机器学习算法可分为两大类:监督学习与无监督学习(图 1)。监督学习算法利用带有标签的训练集来构建数学模型(model),以描述如何通过特征(如基因表达量)的组合与目标变量(如对癌症进展有无影响)进行关联,然后此模型可被用于新数据的预测。标签可以是离散的,比如是否患病,适用于分类算法;也可以是连续的,比如存活率,适用于回归算法。与监督学习相反,无监督学习方法不需要预先标注的数据作为训练集。这一大类算法可以帮助发现或探索数据的结构,其在生物学中广泛应用的例子是数据聚类(clustering),根据观察变量的属性对观察变量进行分组,例如在不同癌症样本中根据基因表达量发现共表达(co-expressed)的基因。
虽然在大数据时代获得数据往往比较容易,但由于获取带标签数据的成本与难度较大,因此大规模数据中往往仅有部分带有标签,而大多数样本是无标签数据,例如大型人群队列检测样本中只有小部分是经诊断的癌症患者样本。此时就需要通过一部分有标签数据对其余数据进行预测。半监督(semi-supervised)学习就是可应用在有标签数据与无标签数据混合的训练数据中的机器学习算法。常见的半监督学习有简单自训练(self-training)、标签传播算法和半监督深度学习等。
2.1 监督学习算法监督学习算法将从带标签的训练集提供的事实中学习,以建立一个通用的模型,用于预测新数据的标签。目前许多著名的监督学习算法(如支持向量机、随机森林、贝叶斯网络)已经应用于微生物组学研究。下面介绍4种常见的监督学习算法(图 2)。

|
| 图 2 四种算法原理图解 Figure 2 Schematic diagram of four algorithms. A: linear regression; B: support vector machine; C: random forest; D: neural network. |
2.1.1 线性回归:
在监督学习方法中,线性回归算法使用线性方程对数据集进行拟合,是一种常见的回归算法。惩罚回归(penalized regression)适用于与样本数量相比包含大量特征的高维数据的回归方法,包括LASSO (least absolute shrinkage and selection operator)、岭回归(ridge regression)和弹性网络。为了避免在变量之间存在相关性时过高估计回归系数,惩罚回归将惩罚函数应用于回归系数。LASSO回归也叫线性回归的L1正则化,该方法最突出的优势在于通过对所有变量系数进行回归惩罚,使得相对不重要的独立变量系数变为0,从而被排除在建模之外。因此,它在拟合模型的同时进行特征选择。岭回归也叫线性回归的L2正则化(平方根函数),它将系数值缩小到接近零,但不删除任何变量。岭回归可以提高预测精准度,但在模型的解释上会更加的复杂化。Zeller等[9]报道了利用LASSO模型与人类肠道微生物组作为CRC (colorectal cancer)筛选工具的可能性,将CRC患者与正常组区别开来。
2.1.2 支持向量机:支持向量机(support vector machine,SVM)是一种二分类模型,它的目的是寻找一个超平面来对样本进行分割,分割的原则是从数据点到超平面的最小距离之和最大化,最终转化为一个凸二次规划问题来求解。一般情况下,当数据分布在二维平面线性可分时,用来确定中间直线(即超平面)只需要虚线上几个点即可(图 2-B),而在三维空间中这些点表现为向量,通过这些向量来支持超平面的生成,距离超平面最近的这些点被称为支持向量。对于更加复杂的数据,往往需要引入核函数,如常用的线性核与高斯核函数,将样本数据从原始空间映射到一个更加高维度的空间,使得样本数据在该空间内是线性可分的。SVM被广泛应用于生物医学多个领域,例如Jie和Robert等[10]引入不同核函数构建SVM分类器,对2702例口腔样本进行分类,探究口腔微生物群落的分类。
2.1.3 随机森林:随机森林(random forest,RF)整合了自举聚合(bootstrap aggregating,bagging)和随机特征选择两种思想。Bagging (装袋)是一种集成学习方法,其使用自助采样法训练多个基础分类器,再将训练好的基础分类器整合起来,获得最终结果。随机森林是Bagging的变体之一,通过随机选择变量的一个子集,并使用每一个子集构建决策树,然后以投票方式集成多棵决策树得到最终决策结果(图 2-C)。随机森林可以用来做分类、回归等问题,也可用于执行特征选择或降维,它被公认为是微阵列分析和其他高维数据领域中表现最好的分类器之一。Yatsunenko等[11]基于肠道菌群16S测序数据,应用随机森林算法对样本进行分类,分析了人类饮食与遗传因素对微生物群落的组成与功能的影响。
2.1.4 人工神经网络与深度学习:人工神经网络(artificial neural network,ANN)最初的设计思想是模仿人脑的工作机制,它是一种图计算模型,被称为神经元(neuron)的计算单元通过分层、相互连接从而相互传递信息(图 2-D)。输入层的所有神经元都连接到第一个隐藏层的所有神经元,每个连接用权值表示。微生物组数据常用OTU矩阵表作为输入层。隐藏层利用反向传播优化输入变量的权值,提高模型的预测能力。这个过程一直持续到最后一个隐藏层连接到输出层为止。输出层是基于来自输入层和隐藏层的数据的预测。深度神经网络或称深度学习(deep learning)指具有多层的神经网络建模技术,通过添加多个隐藏层或层内的节点数,网络可以表现高度复杂的功能。如果给足够大的模型与足够多的带标签的样本,深度学习将能获得优异的性能。目前常见的深度学习框架有TensorFlow (https://www.tensorflow.org)与PyTorch (https://pytorch.org),他们都提供复杂网络结构建模的工具。深度学习已被广泛应用于有监督或无监督(如自动编码器)的机器学习问题,如Lo与Marculescu等[12]利用卷积神经网络(CNN)与微生物组数据预测宿主的疾病状态。
2.2 无监督学习算法与监督学习相反,当标签信息不可用或在建模过程中不使用的情况下称为无监督学习,无监督学习模型可以推断出数据的一些内在结构,主要用于聚类(clustering)与降维(dimension reduction)两个方面。
2.2.1 聚类分析:聚类有助于从已知的数据中探索未知的信息,当样本无标签信息时,基于数据间的相似度将样本分组(簇),划分依据组内距离最小化而组间距离最大化的原则。例如微生物组数据常按16S rDNA序列 > 97%相似度确定为相同物种(species)。常用的聚类方法有K均值(K-means)聚类与层次聚类。聚类最重要的步骤是得到一个好的距离矩阵,图 3列出常见的不同类别之间距离的计算方法。微生物组研究中广泛使用的距离度量是Bray-Curtis和UniFrac差异指标。Bray-Curtis差异指标是量化两个样本之间菌群组成计数的差异,而UniFrac是基于系统发育关系的距离度量指标[7]。对于具有高维特征向量的数据聚类结果,一般通过以下几个指标来评价聚类效果:(1) 聚类中心的距离:距离过大,再分出新类;(2) 聚类域内样本数:聚类数少且离中心点远,考虑为噪音;(3) 聚类域内样本方差:方差过大考虑是否属于该类。
2.2.2 数据降维:
微生物组数据通常都是包含大量OTU变量的高维数据,降维为我们可视化高维数据提供了一种新思路。常用的降维分析方法主要是主成分分析(PCA)、主坐标轴分析(PCoA)和无度量多维尺度法(NMDS)。PCA基于方差分解的原理对多维数据进行降维处理,从而提取出数据中的主要因子和结构,称为主成分(PCs),并以反映数据差异性最大和次大的主成分PC1和PC2用于高维多变量数据的可视化。PCoA与PCA相似,但利用Bray-Curtis或UniFrac距离计算样本间的物种差异度,并选取贡献率最大的主坐标组合进行数据可视化,如果样品距离越接近,表示物种组成越相似。Jiang等[13]基于属水平和OTU水平对植物根际沉积物样本的细菌群落进行PCoA分析,发现细菌群落动态变化主要受根际效应的影响。
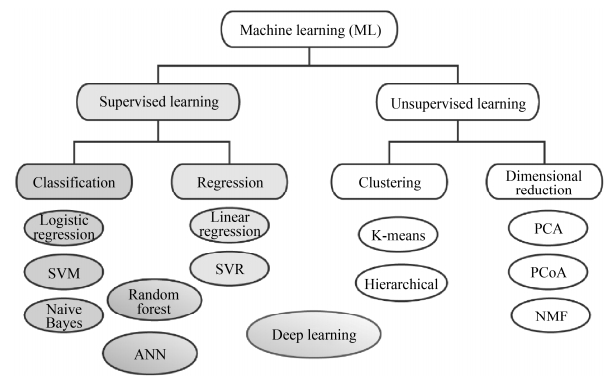
3 机器学习流程在生物学领域,通过机器学习方法进行数据分析的流程主要分为4个方面(图 4):(1) 数据准备(data preparation),包括收集基因序列数据、探索数据及数据预处理,如确定数据是否稀疏及数据缺失程度等,从而确定使用的机器学习算法。(2) 特征工程(feature engineering),对数据进行特征提取、特征选择并将数据分割成训练集与测试集等,准备好数据以输入算法[14]。(3) 模型建立(model generation),采用训练集进行模型训练,并采用测试集对模型进行验证。(4) 模型评估(model evaluation),选择K折交叉验证法(K-fold cross validation)、自助聚合法(bagging)或留一交叉验证法(leave-one-out cross validation,LOOCV)等对模型的性能进行验证。
|
| 图 4 机器学习分析流程 Figure 4 Workflow of machine learning analysis. |
下面介绍基于微生物组数据的机器学习流程中的一些关键要点。
3.1 数据预处理数据预处理主要包括:缺失值处理,即对数据集的错误及缺失数据进行修改或填补;特征提取,即在原始数据中提取更有用的特征或生成新特征;数据降维,用于降低数据的维度,进而降低数据集的复杂度;特征变换,主要包括对数据特征的归一化或标准化等。
在微生物组研究中,根据采集到的样品,获取相关的OTU表数据是微生物组研究的重要步骤[15]。由于微生物组数据的样本量一般较少且OTU特征量较多,导致数据维度大,难以直接进行分析,所以会使用一些减少特征变量的方法,如LASSO、主成分分析等剔除不相关或冗余的特征,从而提高模型准确度。最近Oudah与Henschel提出多层级特征工程(hierarchical feature engineering,HFE)方法,利用OTU特征分层结构的相关性与分类信息进行特征提取,取得更好的预测性能[14]。另外,在大数据样本中,不同属性的量纲往往不一致,数值间的差别也比较大,在机器学习过程中会导致“大数吃小数”等问题。因此,需要对数据进行规范化处理,使之归一化在特定的数值区间,以提高机器学习性能。OTU特征数据一般将每个OTU的值除以每个样本的总读数计算其相对丰度用于机器学习。
3.2 算法选择研究者们提出了各种各样的机器学习算法,算法选择是与预测模型性能优劣有关的重要因素。机器学习算法的选择取决于数据类型、建模目的及应用场景等。没有标签的数据集可用无监督学习算法(如K-means,PCA),而有标签数据集可用监督学习算法(如SVM,RF)。如果标签是类别变量,则可建立分类模型;如果是连续变量,则可建立回归模型。目前多层神经网络的应用日渐成熟,深度学习方法成了热门选择。复杂的模型往往预测性能较好,但模型的可解释性(interpretability)较差[16]。在实际应用中,研究人员希望得到一个可解释性更好的模型用于鉴定与疾病相关的微生物群,而临床医生则希望得到一个预测性能更强的模型。我们通常会选择大家普遍认同的合适算法,如SVM、RF、ANN等。最后,集成方法(ensemble methods)可将多个ML模型整合成一个预测模型,与单个ML模型相比,可以获得更好、更稳健的预测。表 2中总结了一些常见的机器学习算法在预测宿主表型中的应用,更多应用参见https://github.com/LiLabZSU/microbioML中的内容。
| Algorithm | Traita | Samples (P/N) | Performanceb | References |
| SVM | Liver cirrhosis | 118/114 | AUC (0.980) | Pasolli et al., 2016[17] |
| RF | PSC | 24/24 | AUC (0.743) | Iwasawa et al., 2018[18] |
| ANN | Colorectal polyps | 316/236 | AUC (0.870) | Dadkhah et al., 2019[19] |
| Linear regression | IBD | 56/56 | Accuracy (0.780) | Eck et al., 2017[20] |
| Naive bayes | Colorectal polyps | 316/236 | AUC (0.860) | Dadkhah et al., 2019[19] |
| Boosting | IBD | 500/500 | F1-macro (0.650) | Lo et al., 2019[12] |
| K-nearest neighbor | T2D | 423/383 | F1-score (0.860) | Wu et al., 2018[21] |
| Deep learning | Oral malodour | 45/45 | Accuracy (0.967) | Nakano et al., 2018[22] |
| a: PSC: primary sclerosing cholangitis; IBD: inflammatory bowel disease; T2D: diabetes mellitus, type 2. b: The performance metrics were described in the model evaluation section below. | ||||
3.3 数据抽样与交叉验证
机器学习中,通常会将数据集通过重抽样方法(re-sampling)划分为训练集(training set)和测试集(test set)或验证集(validation set)。用于训练模型的数据称为训练集,而对模型进行测试的数据称为测试集。验证集则用于验证模型的泛化性能(generalizability),即可用于其他相似数据的能力。现实中的许多数据的特征存在不平衡的问题,进行机器学习时,测试集数据的分布可能与训练集分布存在差异,将对预测结果产生巨大误差。因此,抽取的样本类别应该大致相等,使得到的样本数据集较为平衡。关于不平衡数据处理方法可参考相关文献[5]。
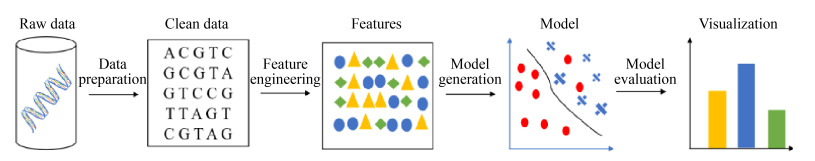
为了充分利用数据集和提高模型的泛化能力,机器学习引入了K-折交叉验证方法。K-折交叉验证(图 5)通过将数据集D分成K个子集,编号D1、D2...Dk,并依次将其中1个子集作为测试集,剩下的K-1个子集作为训练集,使得每一条数据都有均等的几率被选中于训练集和测试集,每个训练集得到预测结果Ek。最后将模型训练和测试后的结果取平均,提高模型的泛化能力。
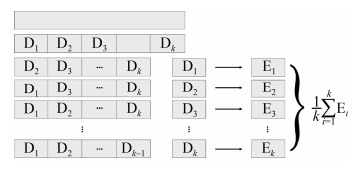
|
| 图 5 K-折交叉验证 Figure 5 K-fold cross validation. |
3.4 模型评估
回归模型的性能评估指标有皮尔斯相关系数(R)、误差平方和(SSE)和均方根误差(RMSE)等。分类模型一般通过计算样本预测值和真实值之间的对应关系来评估模型的性能,评价指标包括预测灵敏度(sensitivity)、精确率(precision)、特异性(specificity)和准确率(accuracy)。灵敏度也称真阳性率或召回率(recall),表示在所有阳性类别的样本中,被正确预测为阳性的比例(公式1);而精确度表示在被预测为阳性的样本中,真正是阳性的比例。特异性也称真阴性率,表示在所有阴性的样本中,被正确预测为阴性的比例(公式2)。准确率表示预测为阳性与阴性样本的总体准确比例(公式3)。

|
公式(1) |

|
公式(2) |

|
公式(3) |
其中,TP:true positive;FN:false negative;TN:true negative;FP:false negative。F1值(F1-score)是精确率和召回率的加权调和平均值(最大值是1,最小值是0)。它有两种计算方式分别为F1-micro与F1-macro,在二分类问题中两者计算方式完全一致,但在多分类问题中有差异。
ROC曲线(receiver operating characteristic curve),又称“受试者工作特性曲线”,最直观的应用就是能反映模型在选取不同阈值的时候其敏感性和特异性的趋势(图 6)。ROC曲线的横坐标轴(1-specificity)表示假阳性率(FPR),即真阴性条件下,预测为阳性;纵坐标表示敏感性(sensitivity)或真阳性率(TPR)。根据ROC曲线位置,曲线下方部分的面积被称为AUC (area under curve),用来表示预测准确性,AUC值越高,说明预测准确率越高,即曲线越接近左上角,预测准确率越高。一般AUC在0.5到1.0之间,当AUC < 0.5时,说明模型没有意义。Ai等[23]通过AUC显示不同模型的预测效果,得出Bayes和随机森林模型具有较好的预测能力。
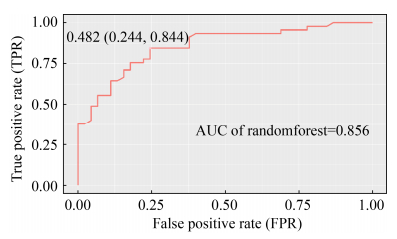
|
| 图 6 口臭和健康呼吸分类的ROC曲线 Figure 6 ROC curve for classification of malodourous and healthy breath. |
4 案例分析
本案例来自Yoshio等[22]通过机器学习基于口腔微生物群进行口腔异味预测的研究,基于90例受试者唾液样本的16S rRNA基因扩增子数据分析所得到的OTU矩阵,对口腔异味和健康呼吸进行机器学习分类预测。本例将通过开源免费软件R中的随机森林、支持向量机等机器学习算法得到分类预测模型,并绘制ROC曲线以评价各算法的预测性能。
4.1 硬件与软件 4.1.1 硬件:运行Windows、Linux或macOS操作系统的64位计算机;计算机的处理器与内存需求依赖分析数据的大小与分析模型。
4.1.2 软件:使用开源免费软件R (https://www.r-roject.org),想要了解更多基于R实现的机器学习库,可以参考CRAN中机器学习的任务视图(https://cran.r-project.org/web/views/MachineLearning.html)。
通过以下命令安装本案例需要的R包:install.packages (c(“randomForest”,“pROC”,“ggplot2”))。
4.2 数据集(dataset)Yoshio等使用的研究对象包括90名患者(男性37名,女性53名,平均年龄50.0±14.7岁),他们于2011年8月至2016年10月期间在福冈牙科医学院和牙科医院口腔异味诊所就诊,主述口臭。3个月内未服用抗生素,无耳鼻喉或代谢性疾病。在这90位病人中,45位没有或有微弱的口腔异味,45位有明显的口腔异味。Nakano Y等[22]的文章中文件12903_2018_591_MOSESM1_ESM.csv (此处重命名S1_table.csv)是一个90×109的OTU数据集(https://github.com/LiLabZSU/microbioML),第二列“Malodour”表示呼吸为有口臭(P,阳性)或正常(N,阴性)。
4.3 建立模型(model)下面介绍如何在R中基于微生物组数据构建口腔异味预测模型的分析过程。关于模型构建更详细代码参考https://github.com/LiLabZSU/microbioML中的内容。
4.3.1 加载数据:在R中设置本机工作目录在“D: \microbioML”,并载入所需要的数据集:S1_table.csv(样本中OTU丰度数据集)。
setwd(“D: \microbioML”) # 按数据实际路径修改
data < - read.csv(“S1_table.csv”, header=TRUE, row.names=1, sep=“, ”)
data$Malodour < -as.factor(data$Malodour)
head(data) #显示部分数据
4.3.2 数据分次交叉验证:进行交叉检验首先要对数据分组,数据分组要符合随机且平均的原则。K-折交叉验证(K-fold cross-validation)是交叉验证方法里一种,数据分折(k-fold)函数如下:
CVgroup < - function(k, datasize, seed){
cvlist < - data.frame()
set.seed(seed)
n < - rep(1:k, ceiling(datasize/k))[1:datasize] #将数据分成k份,并生成的完整数据集n
temp < - sample(n, datasize) #随机化
x < - 1:k
dataseq < - 1:datasize
cvlist < - lapply(x, function(x) dataseq[temp == x]) #随机生成k个随机有序数据列
return(cvlist)
} #定义分折函数
k < - 10 #k-fold设为10
datasize < - 90 #样本数为90
cvlist < - CVgroup(k = k, datasize = datasize, seed = 123) #代入参数
4.3.3 随机森林模型:随机森林预测模型使用R包randomForest构建,变量pred显示实际值与预测初步结果概率的数据框,方便绘制ROC曲线。
library(randomForest) #载入模型库
pred < - data.frame() #存储预测结果
for (i in 1:10) {
train < - data[-cvlist[[i]], ] #训练集
test < - data[cvlist[[i]], ] #测试集
#建立randomforest模型,ntree指定树数
rf.model < - randomForest(Malodour ~., data = train, ntree=100)
summary(rf.model)
rf.pred < - predict(rf.model, test, type ="prob")[, 2] #预测
kcross < - rep(i, length(rf.pred)) #i为第几次循环交叉,共K次
temp < - cbind(Malodour = test$Malodour, rfPredict = as.data.frame(rf.pred), kcross)
pred < - rbind(pred, temp)
}#循环计算K次模型
head(pred)
4.3.4 绘制ROC曲线:通过pROC包里的roc函数和ggplot2里的ggroc函数绘制ROC曲线。
library(pROC)
library(ggplot2)
rf.roc < - roc(pred $ Malodour, as.numeric (pred $ rf.pred))
rf.roc$auc
ggroc(rf.roc, alpha=0.5, colour=“red”, linetype=1, size=2, legacy.axes = TRUE) +
annotate(“text”, x =.75, y =.15, label = paste(“AUC of Random Forest =”, round (rf.roc$auc, 3)))
4.4 模型性能评价与文献[22]中报道的SVM机器学习结果类似,本研究对唾液样本的16S rRNA基因扩增子测序所得的微生物组成数据进行随机森林分类器预测的正确率(accuracy)也为80%左右(图 6)。
ROC曲线显示随着真阳性率(sensitivity)的上升,假阳性率(1-specificity)也相应增加。最接近ROC曲线左上角的那一点为最佳临界点(0.244,0.844),对应分类阈值是0.482,模型的灵敏度和特异度达到较好平衡。ROC曲线下面积(AUC)体现模型准确性,随机森林模型的AUC值为0.856,说明模型预测准确性较高,即当输入一组新样本数据时可以较好地判断出样本来源受试者是否有口腔异味。
5 展望微生物对其宿主表型的影响往往不是通过某一两种细菌来决定,而是通过微生物群的协同作用来决定。因此,一旦我们确定了所研究宿主的微生物组特征,可以使用机器学习方法来探索微生物组与宿主表型之间的相互作用关系。微生物组机器学习研究一般首先以16S rRNA基因序列聚类后的OTU表作为微生物概况数据,进行数据预处理与特征选择;然后使用合适的机器学习算法构建预测模型,并利用测试数据集评估模型性能。虽然机器学习等人工智能方法已成功应用于生物医学领域,但目前还面临许多问题,如模型的精度有待提高、泛化能力还需加强等,尤其微生物组数据往往存在研究样本量少、特征多导致过拟合的问题。为了应对这些挑战,机器学习未来还需要在特征工程与算法等方面有所改进。目前大多微生物组研究基于16S rRNA基因测序数据,而宏基因组测序(shotgun metagenomic)数据能提供菌种(species)或菌株(strains)水平的分类信息[24],并可采用K-mers代替OTUs作为特征,由于K-mers分析不需要序列比对,将大大加快分析速度。另外,随着大样本队列微生物组研究的增加,深度学习算法将能应用于构建预测模型,而对于大样本量数据(如 > 1万例),深度学习会有更精确的预测结果[6]。最后,为衡量ML算法模型的可靠性,还需一个由全球科学社区共同建设并维护的公共微生物组数据库,如Microbiome Learning Repo (ML-repo)[25]。总之,随着基因组测序与人工智能技术的快速发展,机器学习将成为基于微生物组数据预测宿主性状或疾病诊断的重要工具。
| [1] | Dewhirst FE, Chen T, Izard J, Paster BJ, Tanner ACR, Yu WH, Lakshmanan A, Wade WG. The human oral microbiome. Journal of Bacteriology, 2010, 192(19): 5002-5017. DOI:10.1128/JB.00542-10 |
| [2] | Cammarota G, Ianiro G, Ahern A, Carbone C, Temko A, Claesson MJ, Gasbarrini A, Tortora G. Gut microbiome, big data and machine learning to promote precision medicine for cancer. Nature Reviews Gastroenterology & Hepatology, 2020, 17(10): 635-648. |
| [3] |
Zhang GQ, Huang ZQ, Wang MY, Kong JH, Li YD, Chen JS. Association between dietary habits and salivary microbial diversity in college students. Food Science, 2019, 40(1): 196-201.
(in Chinese) 张国庆, 黄子琪, 王明月, 孔俊豪, 李余动, 陈建设. 大学生饮食习惯与唾液微生物多样性的关联. 食品科学, 2019, 40(1): 196-201. |
| [4] | Mingle D. Machine learning techniques on microbiome-based diagnostics. Advances in Biotechnology & Microbiology, 2017, 6(4): 97-99. |
| [5] | Zhou YH, Gallins P. A review and tutorial of machine learning methods for microbiome host trait prediction. Frontiers in Genetics, 2019, 10: 579. DOI:10.3389/fgene.2019.00579 |
| [6] | Li Y, Huang C, Ding LZ, Li ZX, Pan YJ, Gao X. Deep learning in bioinformatics: Introduction, application, and perspective in the big data era. Methods, 2019, 166: 4-21. DOI:10.1016/j.ymeth.2019.04.008 |
| [7] | Knight R, Vrbanac A, Taylor BC, Aksenov A, Callewaert C, Debelius J, Gonzalez A, Kosciolek T, McCall LI, McDonald D, Melnik AV, Morton JT, Navas J, Quinn RA, Sanders JG, Swafford AD, Thompson LR, Tripathi A, Xu ZZ, Zaneveld JR, Zhu QY, Caporaso JG, Dorrestein PC. Best practices for analysing microbiomes. Nature Reviews Microbiology, 2018, 16(7): 410-422. DOI:10.1038/s41579-018-0029-9 |
| [8] | Zitnik M, Nguyen F, Wang B, Leskovec J, Goldenberg A, Hoffman MM. Machine learning for integrating data in biology and medicine: Principles, practice, and opportunities. Information Fusion, 2019, 50: 71-91. |
| [9] | Zeller G, Tap J, Voigt AY, Sunagawa S, Kultima JR, Costea PI, Amiot A, Böhm J, Brunetti F, Habermann N, Hercog R, Koch M, Luciani A, Mende DR, Schneider MA, Schrotz-King P, Tournigand C, Tran van Nhieu J, Yamada T, Zimmermann J, Benes V, Kloor M, Ulrich CM, von Knebel Doeberitz M, Sobhani I, Bork P. Potential of fecal microbiota for early-stage detection of colorectal cancer. Molecular Systems Biology, 2014, 10: 766. DOI:10.15252/msb.20145645 |
| [10] | Ning J, Beiko RG. Phylogenetic approaches to microbial community classification. Microbiome, 2015, 3(1): 1-13. DOI:10.1186/s40168-014-0066-1 |
| [11] | Yatsunenko T, Rey FE, Manary MJ, Trehan I, Dominguez-Bello MG, Contreras M, Magris M, Hidalgo G, Baldassano RN, Anokhin AP, Heath AC, Warner B, Reeder J, Kuczynski J, Gregory Caporaso J, Lozupone CA, Lauber C, Clemente JC, Knights D, Knight R, Gordon JI. Human gut microbiome viewed across age and geography. Nature, 2012, 486(7402): 222-227. DOI:10.1038/nature11053 |
| [12] | Lo C, Marculescu R. MetaNN: accurate classification of host phenotypes from metagenomic data using neural networks. BMC Bioinformatics, 2019, 20(Suppl 12): 314. DOI:10.1186/s12859-019-2833-2 |
| [13] | Jiang XT, Peng X, Deng GH, Sheng HF, Wang Y, Zhou HW, Tam NFY. Illumina sequencing of 16S rRNA tag revealed spatial variations of bacterial communities in a mangrove wetland. Microbial Ecology, 2013, 66(1): 96-104. DOI:10.1007/s00248-013-0238-8 |
| [14] | Oudah M, Henschel A. Taxonomy-aware feature engineering for microbiome classification. BMC Bioinformatics, 2018, 19(1): 227. DOI:10.1186/s12859-018-2205-3 |
| [15] | Namkung J. Machine learning methods for microbiome studies. Journal of Microbiology, 2020, 58(3): 206-216. DOI:10.1007/s12275-020-0066-8 |
| [16] | Topçuoğlu B, Lesniak N, Ruffin M, Wiens J, Schloss P. A Framework for Effective Application of Machine Learning to Microbiome-Based Classification Problems. mBio, 2020, 11(3): 1-13. |
| [17] | Pasolli E, Truong DT, Malik F, Waldron L, Segata N. Machine learning meta-analysis of large metagenomic datasets: tools and biological insights. PLoS Computational Biology, 2016, 12(7): e1004977. DOI:10.1371/journal.pcbi.1004977 |
| [18] | Iwasawa K, Suda W, Tsunoda T, Oikawa-Kawamoto M, Umetsu S, Takayasu L, Inui A, Fujisawa T, Morita H, Sogo T, Hattori M. Dysbiosis of the salivary microbiota in pediatric-onset primary sclerosing cholangitis and its potential as a biomarker. Scientific Reports, 2018, 8: 5480. DOI:10.1038/s41598-018-23870-w |
| [19] | Dadkhah E, Sikaroodi M, Korman L, Hardi R, Baybick J, Hanzel D, Kuehn G, Kuehn T, Gillevet PM. Gut microbiome identifies risk for colorectal polyps. BMJ Open Gastroenterology, 2019, 6(1): e000297. DOI:10.1136/bmjgast-2019-000297 |
| [20] | Eck A, Zintgraf LM, de Groot EFJ, de Meij TGJ, Cohen TS, Savelkoul PHM, Welling M, Budding AE. Interpretation of microbiota-based diagnostics by explaining individual classifier decisions. BMC Bioinformatics, 2017, 18(1): 1-13. |
| [21] | Wu HL, Cai LH, Dongfang L, Wang XY, Zhao SC, Zou FH, Zhou K. Metagenomics biomarkers selected for prediction of three different diseases in Chinese population. BioMed Research International, 2018, 2018: 2936257. |
| [22] | Nakano Y, Suzuki N, Kuwata F. Predicting oral malodour based on the microbiota in saliva samples using a deep learning approach. BMC Oral Health, 2018, 18(1): 128. DOI:10.1186/s12903-018-0591-6 |
| [23] | Ai LY, Tian HY, Chen ZF, Chen HM, Xu J, Fang JY. Systematic evaluation of supervised classifiers for fecal microbiota-based prediction of colorectal cancer. Oncotarget, 2017, 8(6): 9546-9556. DOI:10.18632/oncotarget.14488 |
| [24] | Carrieri AP, Rowe WP, Winn M, Pyzer-Knapp EO. A fast machine learning workflow for rapid phenotype prediction from whole shotgun metagenomes. Proceedings of the AAAI Conference on Artificial Intelligence, 2019, 33: 9434-9439. |
| [25] | Vangay P, Hillmann BM, Knights D. Microbiome Learning Repo (ML Repo): a public repository of microbiome regression and classification tasks. Giga Science, 2019, 8(5): giz042. |
 2021, Vol. 61
2021, Vol. 61